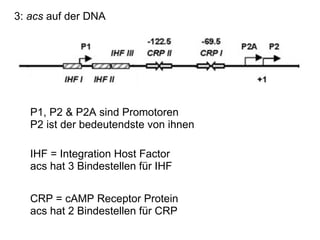
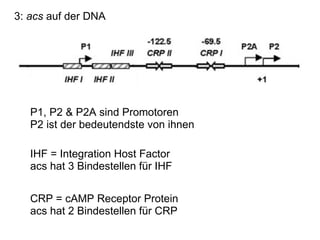
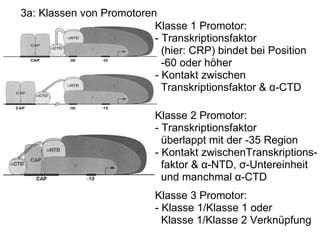
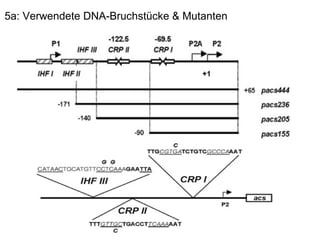
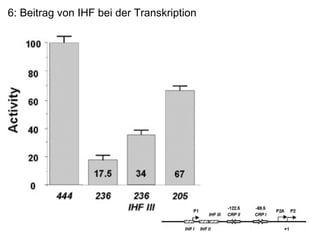
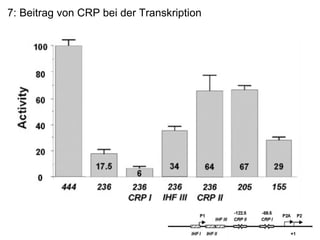
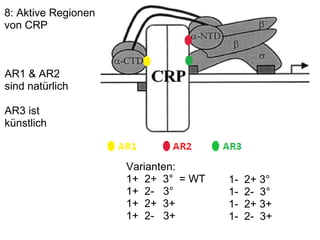
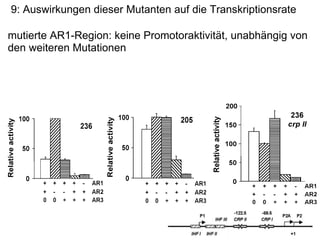
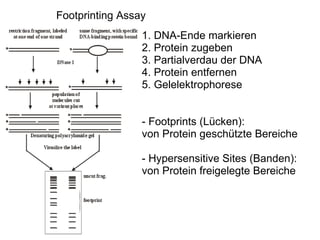
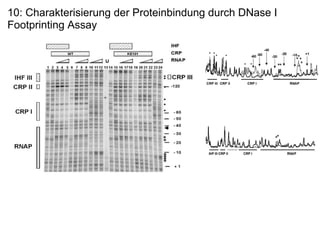
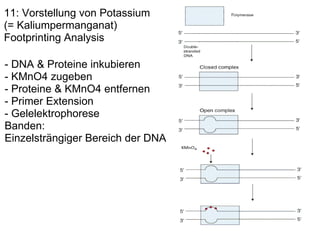
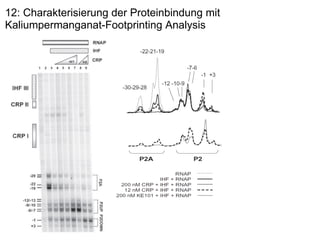
Das Dokument untersucht die Rolle des acs-Gens, das für die Acetyl-CoA-Synthetase verantwortlich ist, und die Wechselwirkungen zwischen den Proteinen IHF und CRP an den Promotoren P1, P2 und P2a, wobei P2 der bedeutendste ist. Die Aktivierung des Promotors erfolgt bei Kohlenstoffmangel durch Bindung von CRP und IHF, die die RNA-Polymerase rekrutieren, um die Transkription zu starten. Verschiedene Mutationen in den aktiven Regionen von CRP beeinflussen die Transkriptionsrate, wobei das Dokument verschiedene Methoden zur Analyse der Protein-DNA-Interaktionen beschreibt.