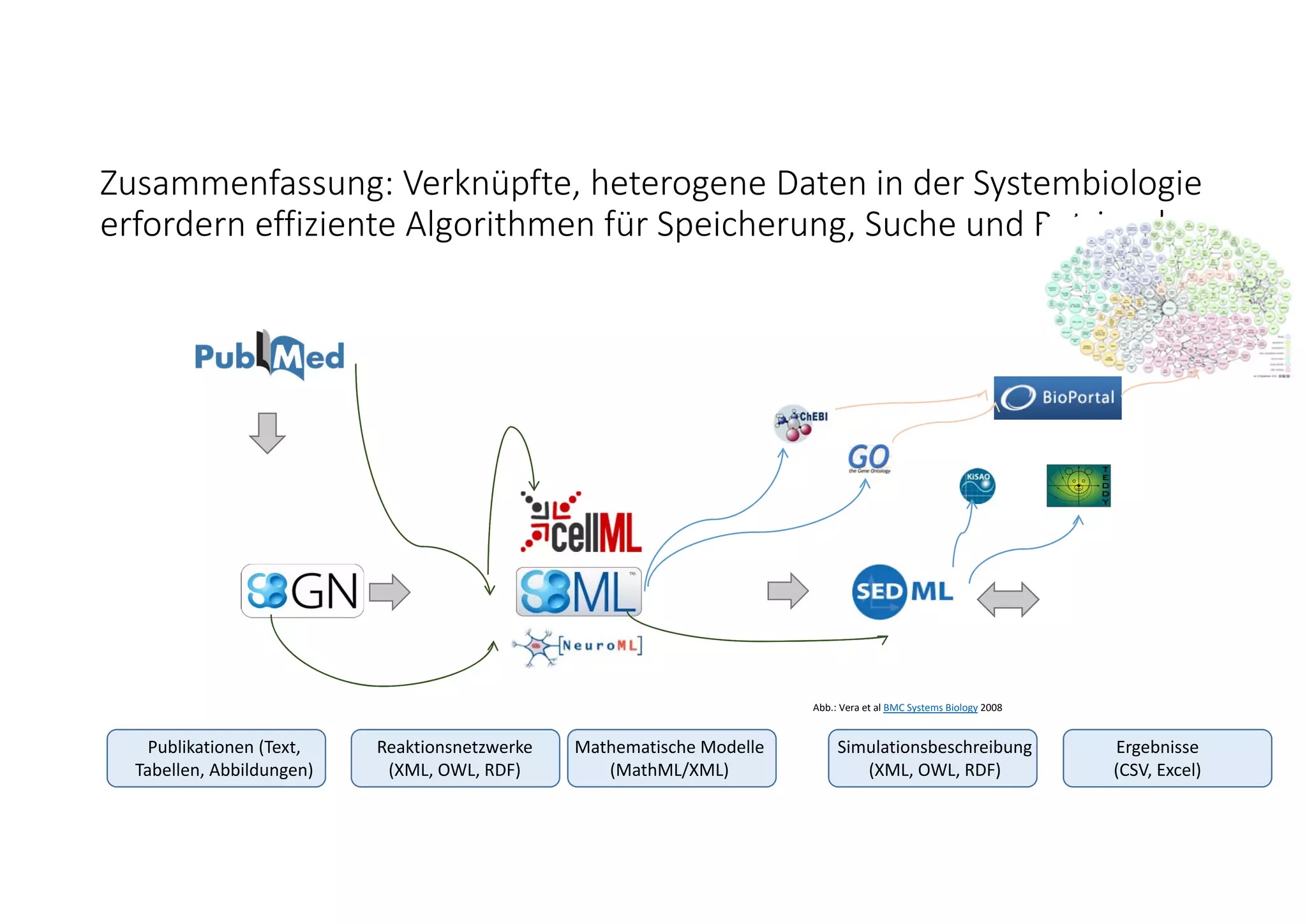
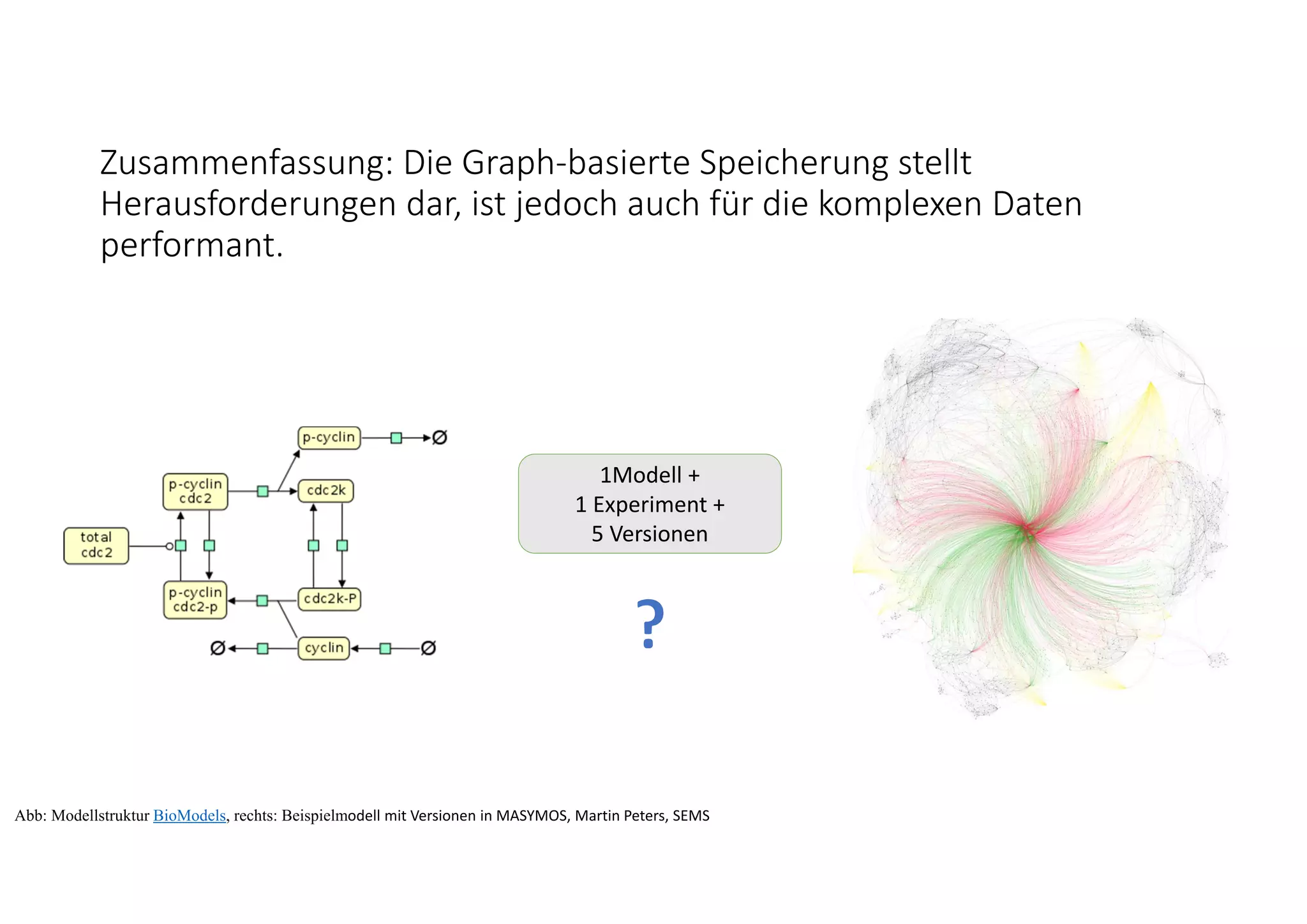
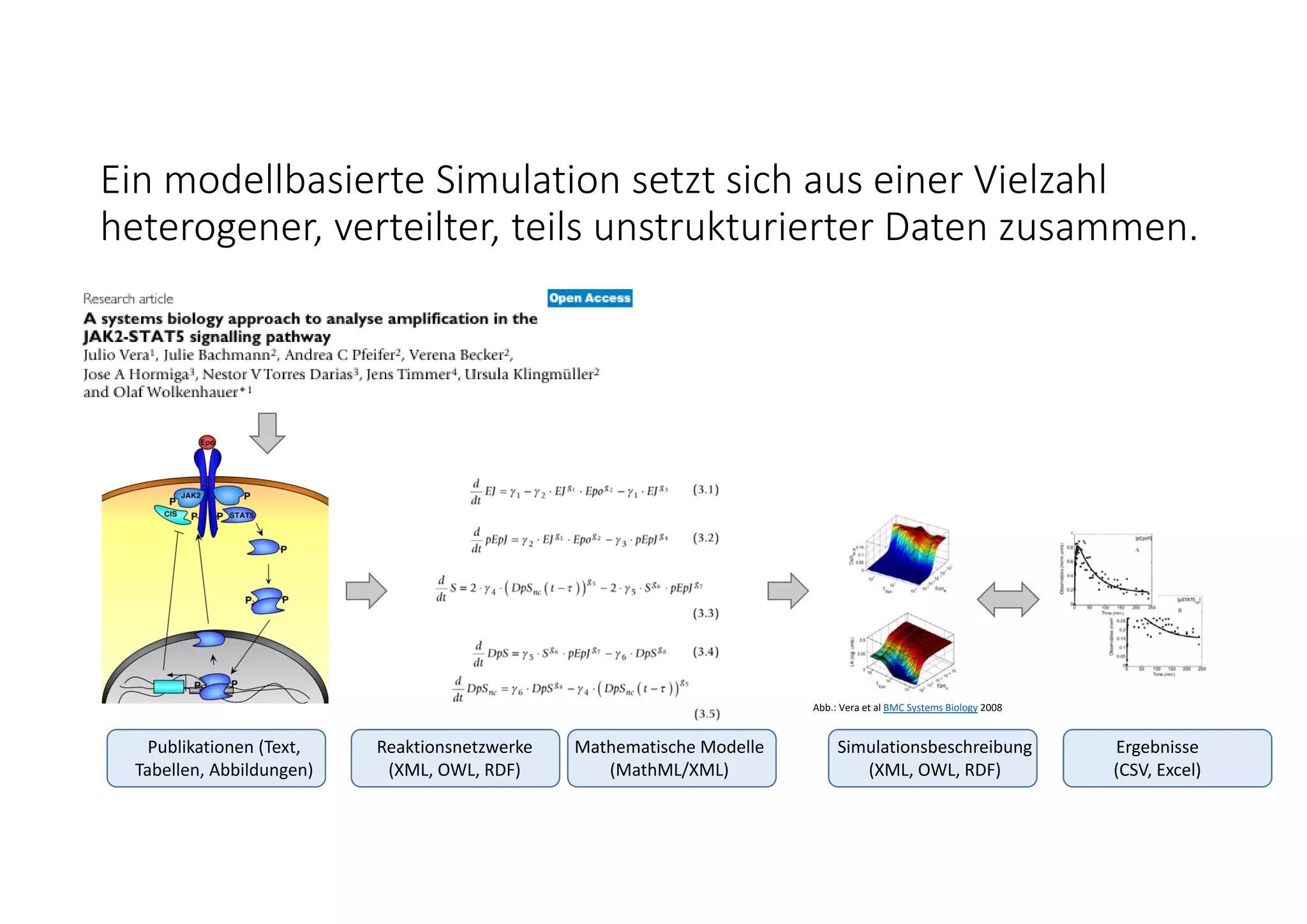
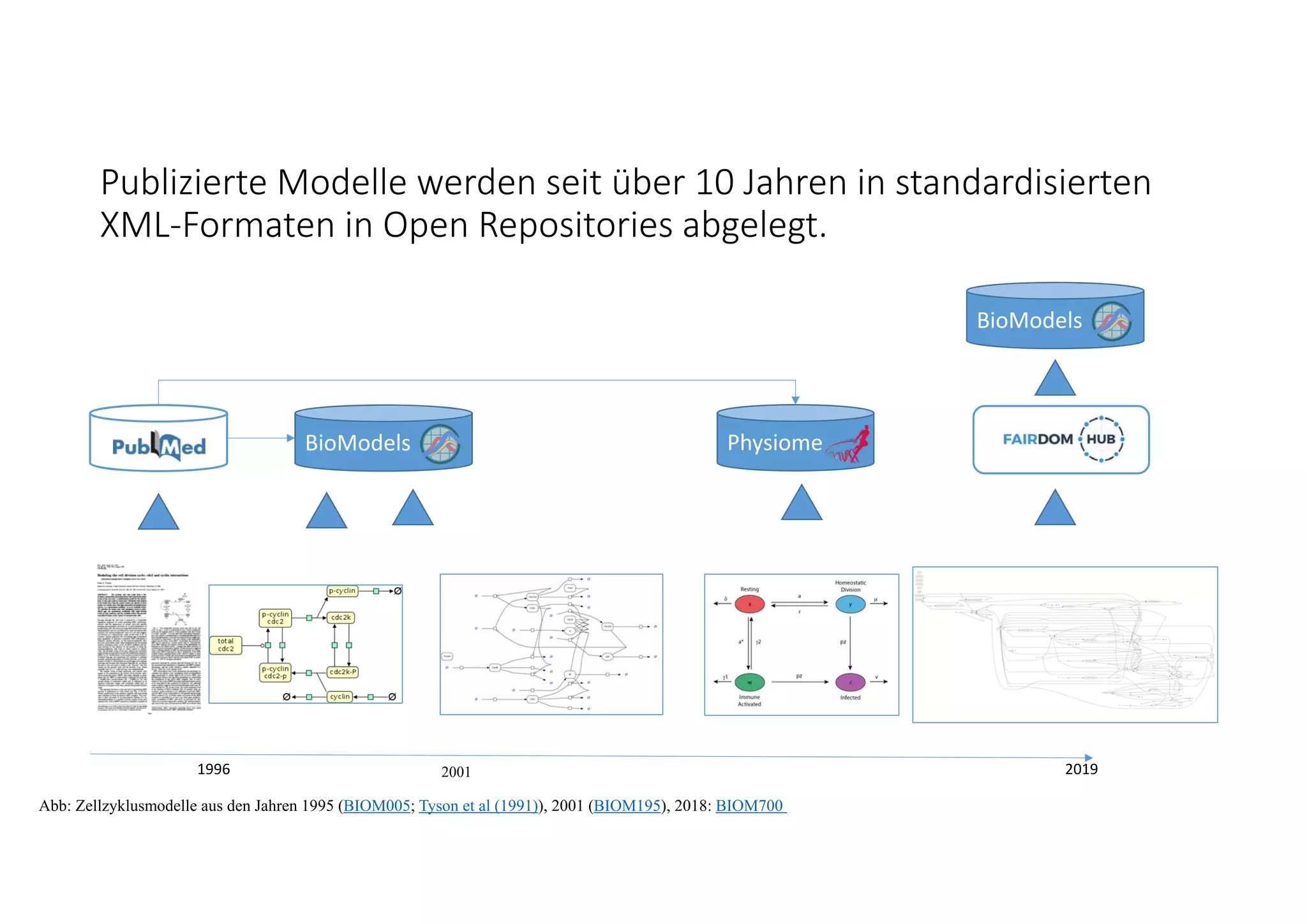
Das Dokument beschreibt graph-basierte Ansätze zur effizienten Speicherung und Verwaltung von heterogenen Daten in der Systembiologie, insbesondere durch den Einsatz von Neo4j und Lucene für die Modellspeicherung und den Abruf. Es werden Herausforderungen und Lösungen im Bereich der Reproduzierbarkeit, Integration und Modellentwicklung dargelegt, sowie Entwicklungen in der Modellversionierung und Strukturähnlichkeitssuche aufgezeigt. Die Autoren betonen die Notwendigkeit flexibler Anfragesysteme für die zunehmende Komplexität und Anzahl von biologischen Modellsimulationen.
![[XML+RelDB] [GraphDB+Cypher+Lucene] = max(efficiency|flexibility)
oder
Implementierung Graph‐basierter Ansätze
für das Management systembiologischer
Simulationsmodelle
Dagmar Waltemath
Medizininformatik
Universitätsmedizin Greifswald
ORCID: 0000‐0002‐5886‐5563
Twitter: @dagmarwaltemath
Ron Henkel
Business Information Systems
Universität Rostock
ORCID: 0000‐0001‐6211‐2719
E‐Mail: ron.henkel@uni‐rostock.de
Web‐Meeting German Biobank Alliance | 10.05.2019](https://image.slidesharecdn.com/waltemath-henkel-19-05-10-germanbiobank-190511201744/75/Implementierung-Graph-basierter-Ansatze-fur-das-Management-systembiologischer-Simulationsmodelle-1-2048.jpg)







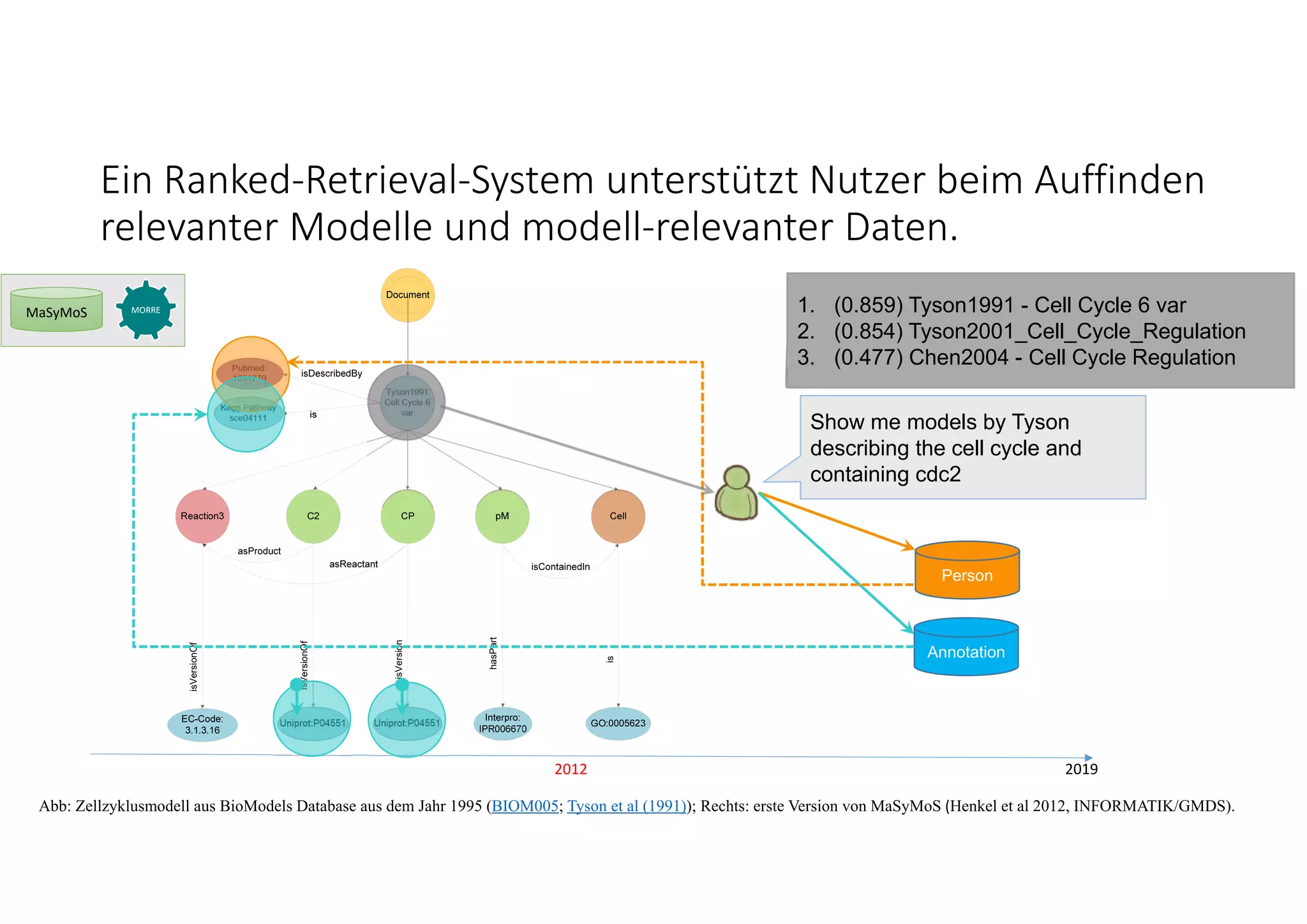
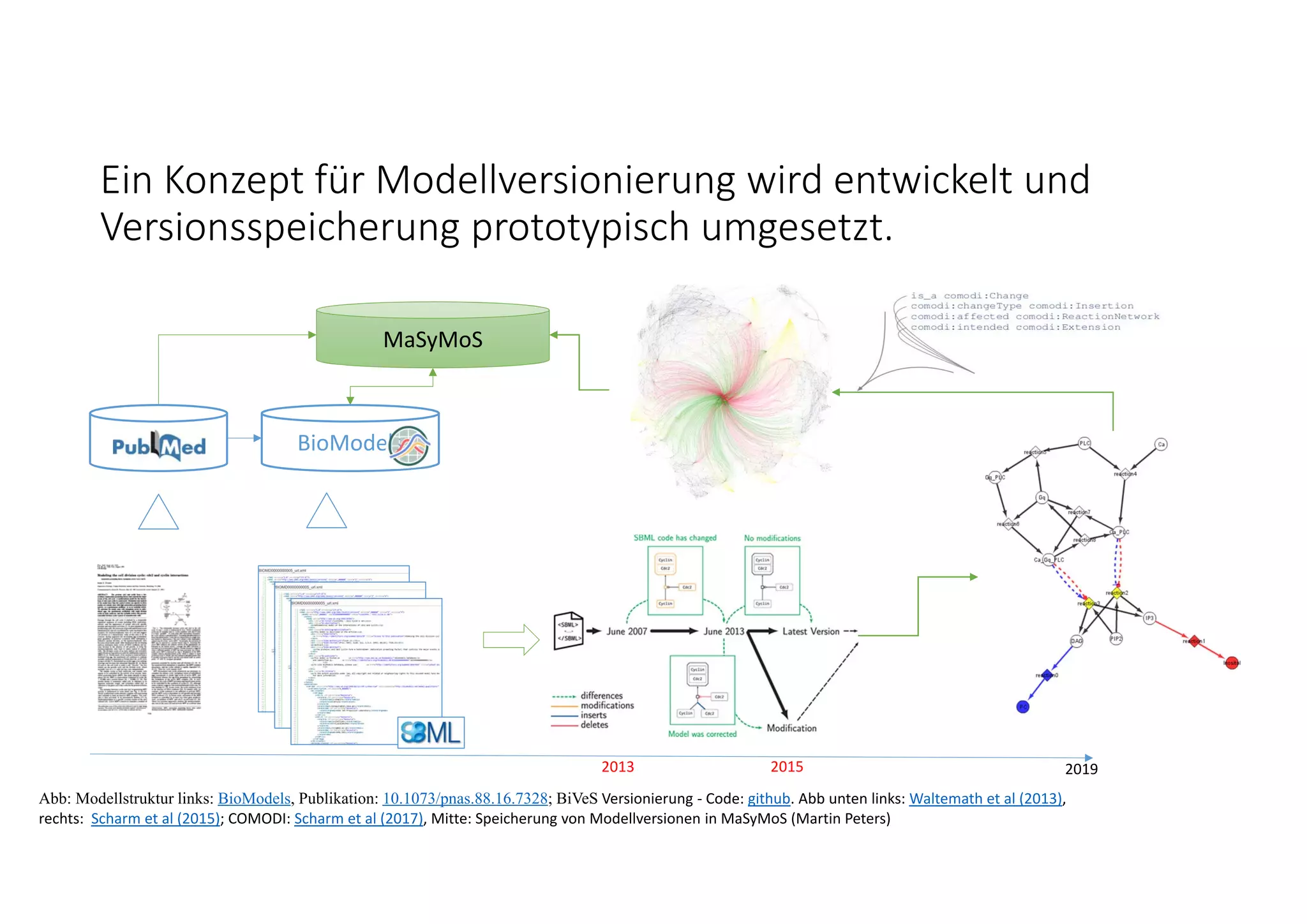
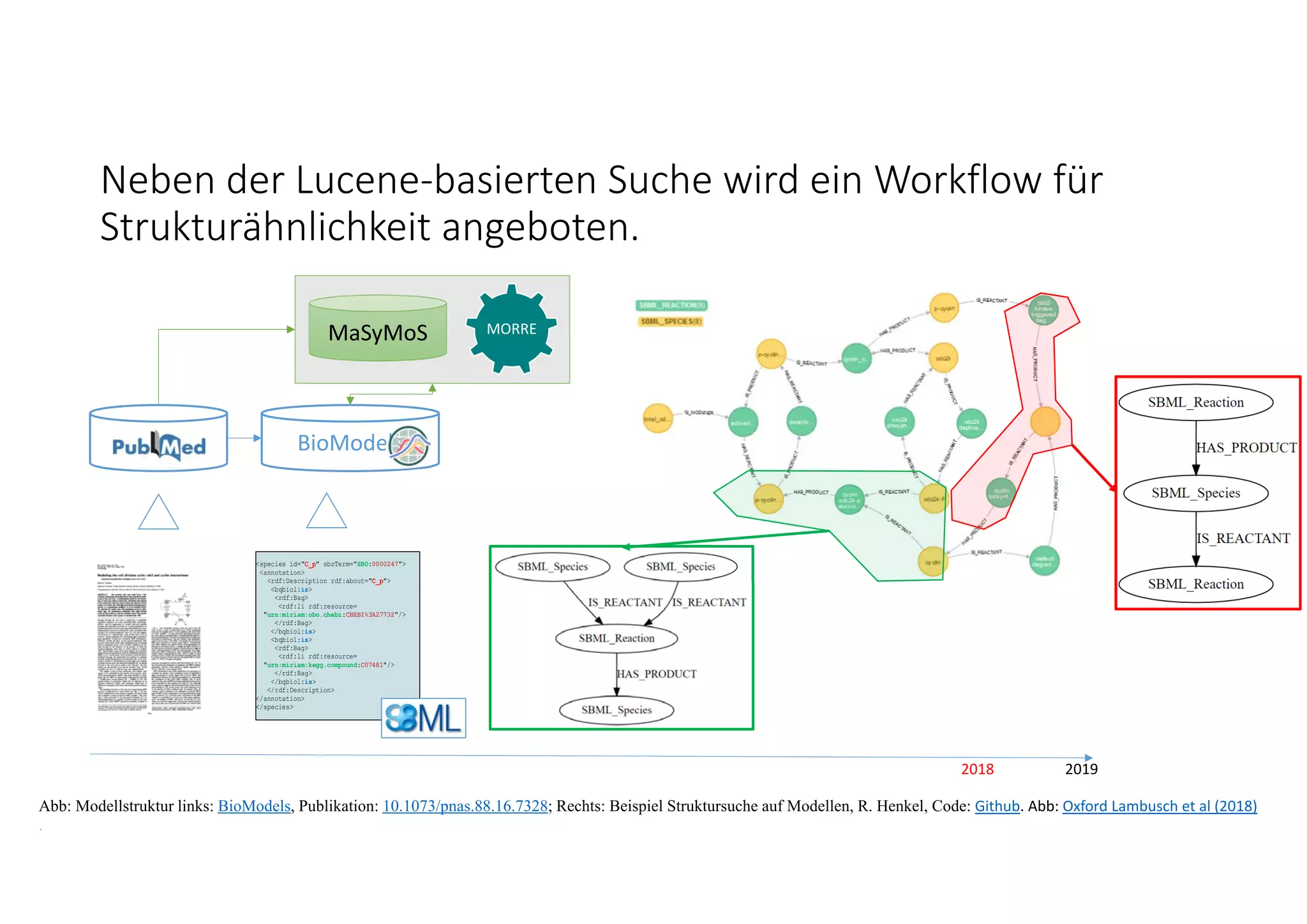
![Neben der Lucene‐basierten Suche wird ein Workflow für
Strukturähnlichkeit angeboten.
2018 2019
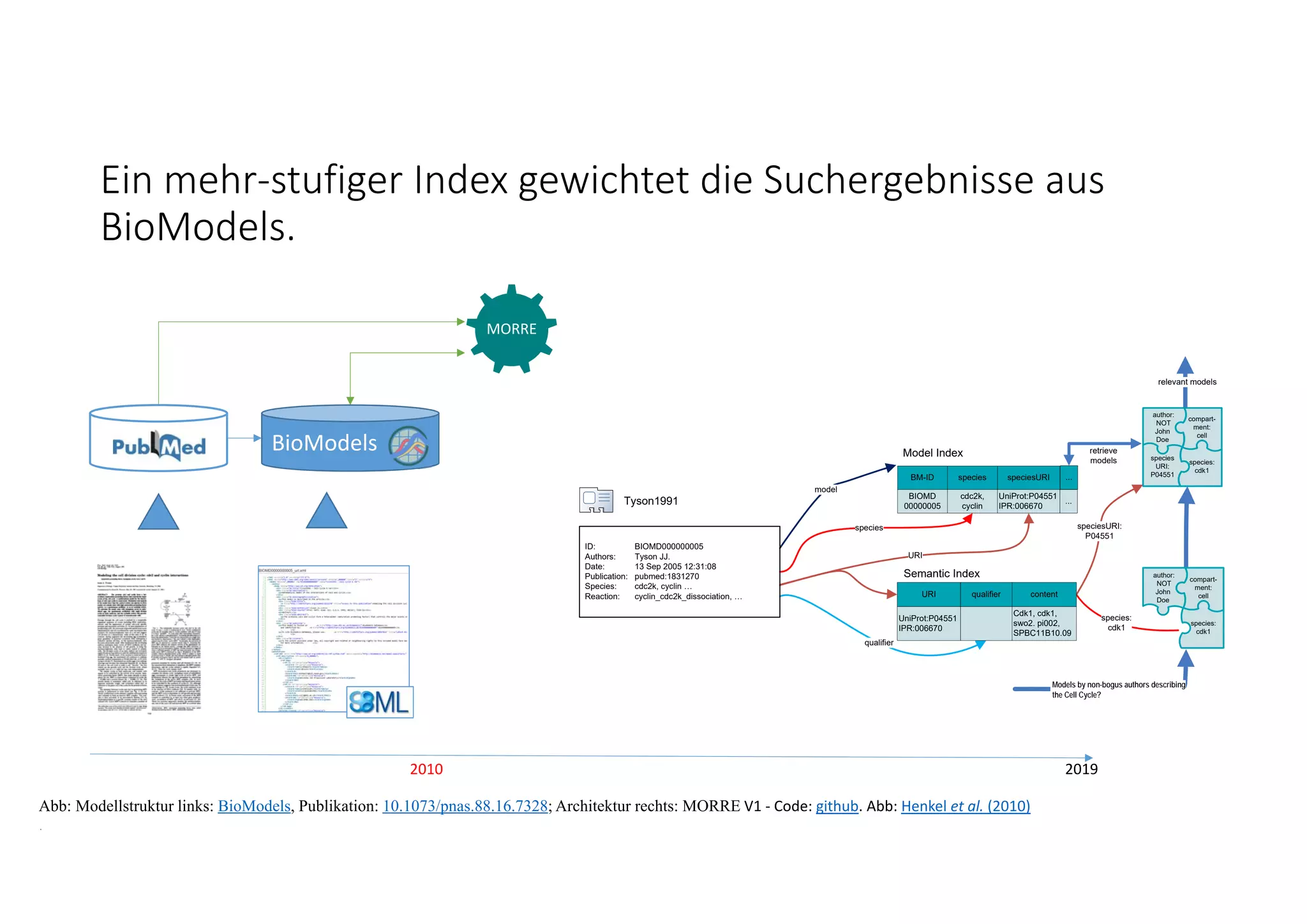
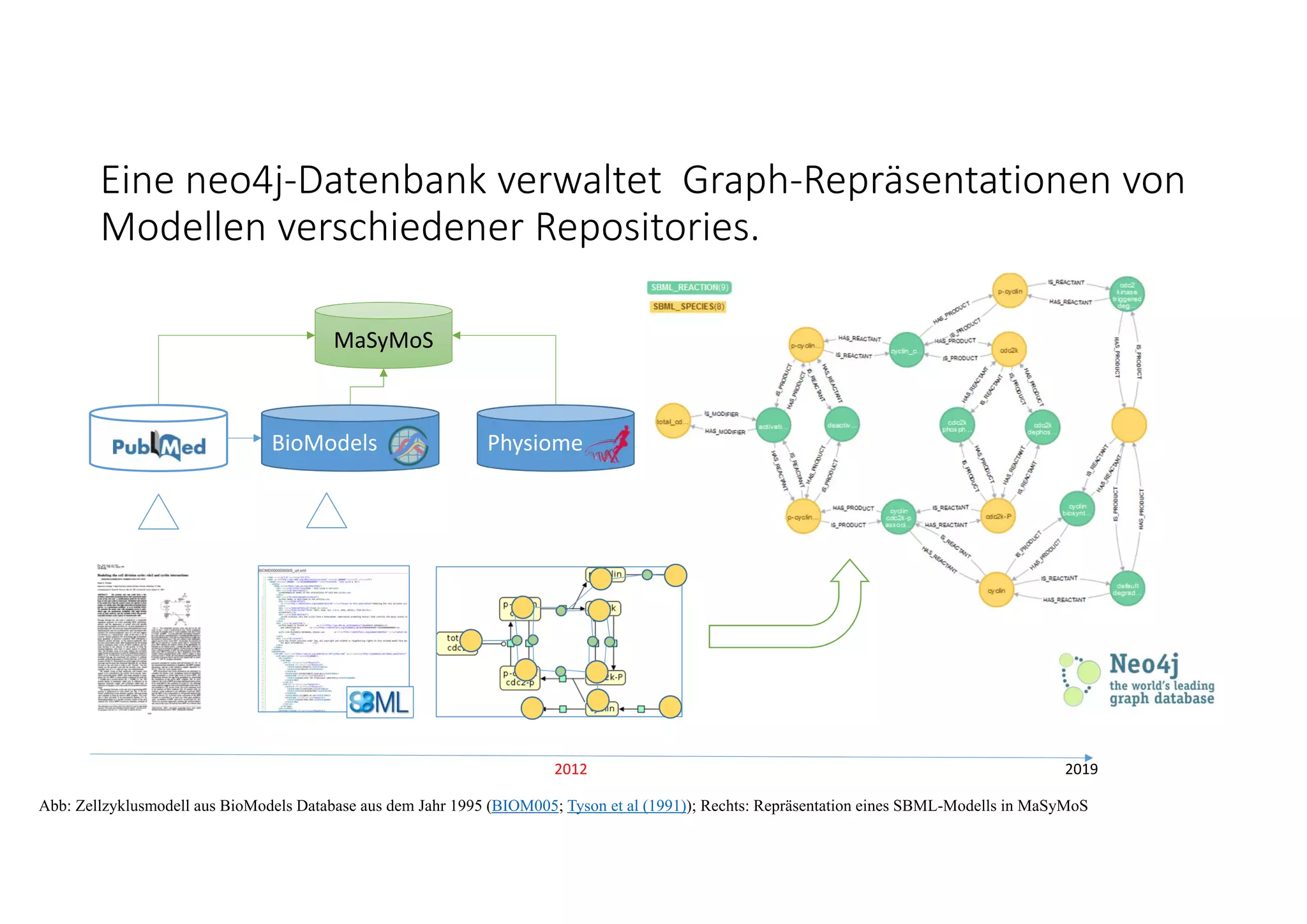
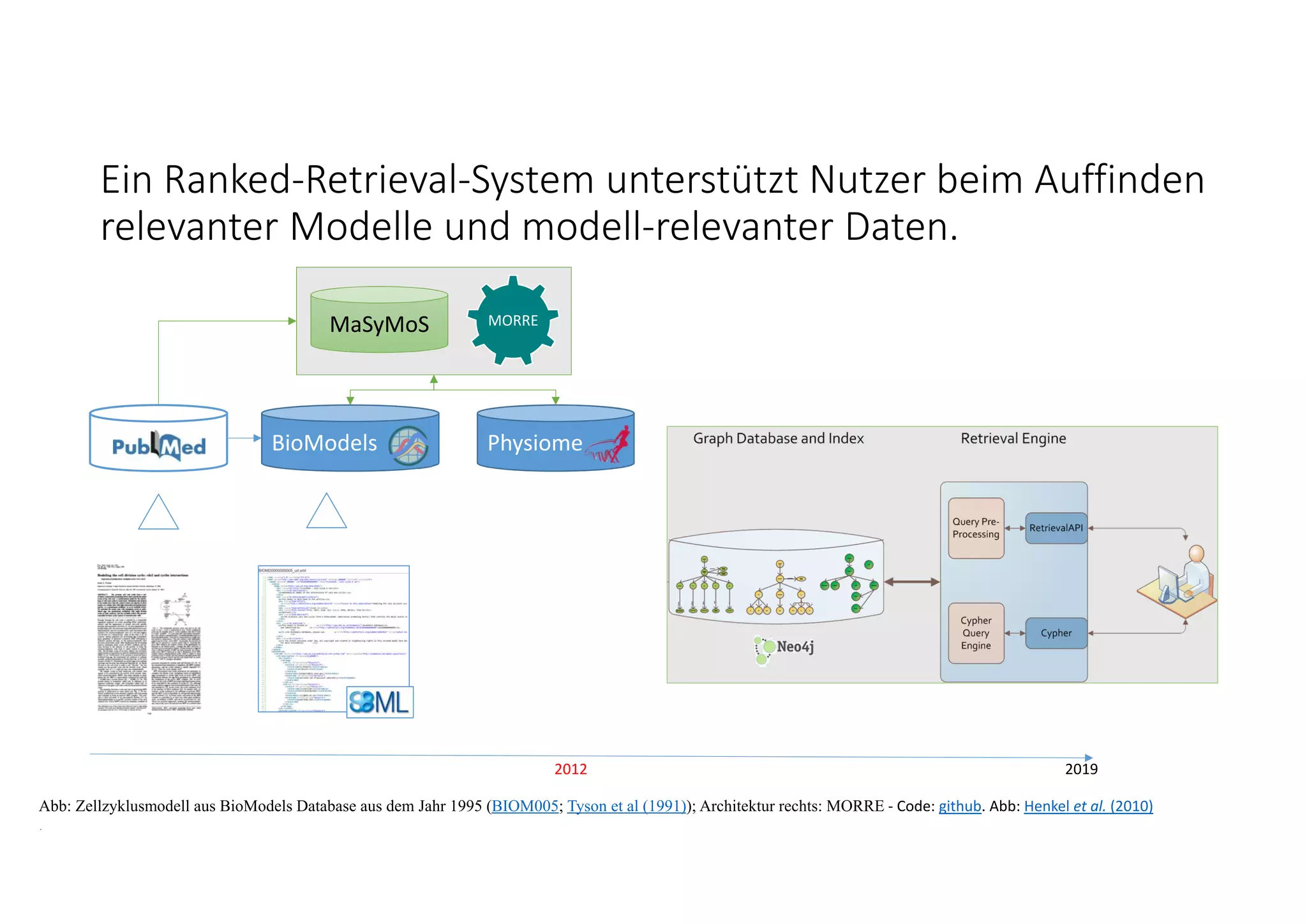
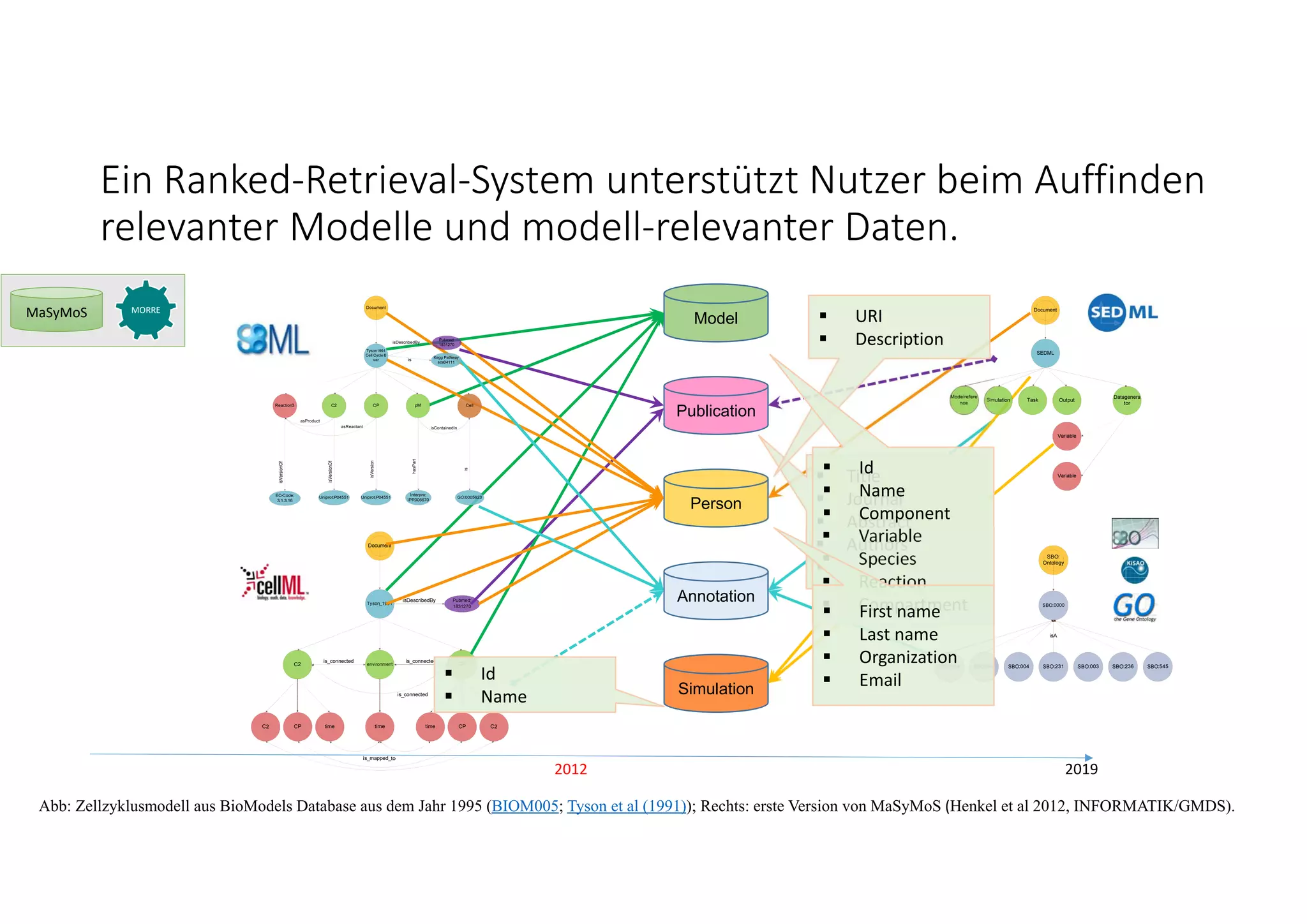
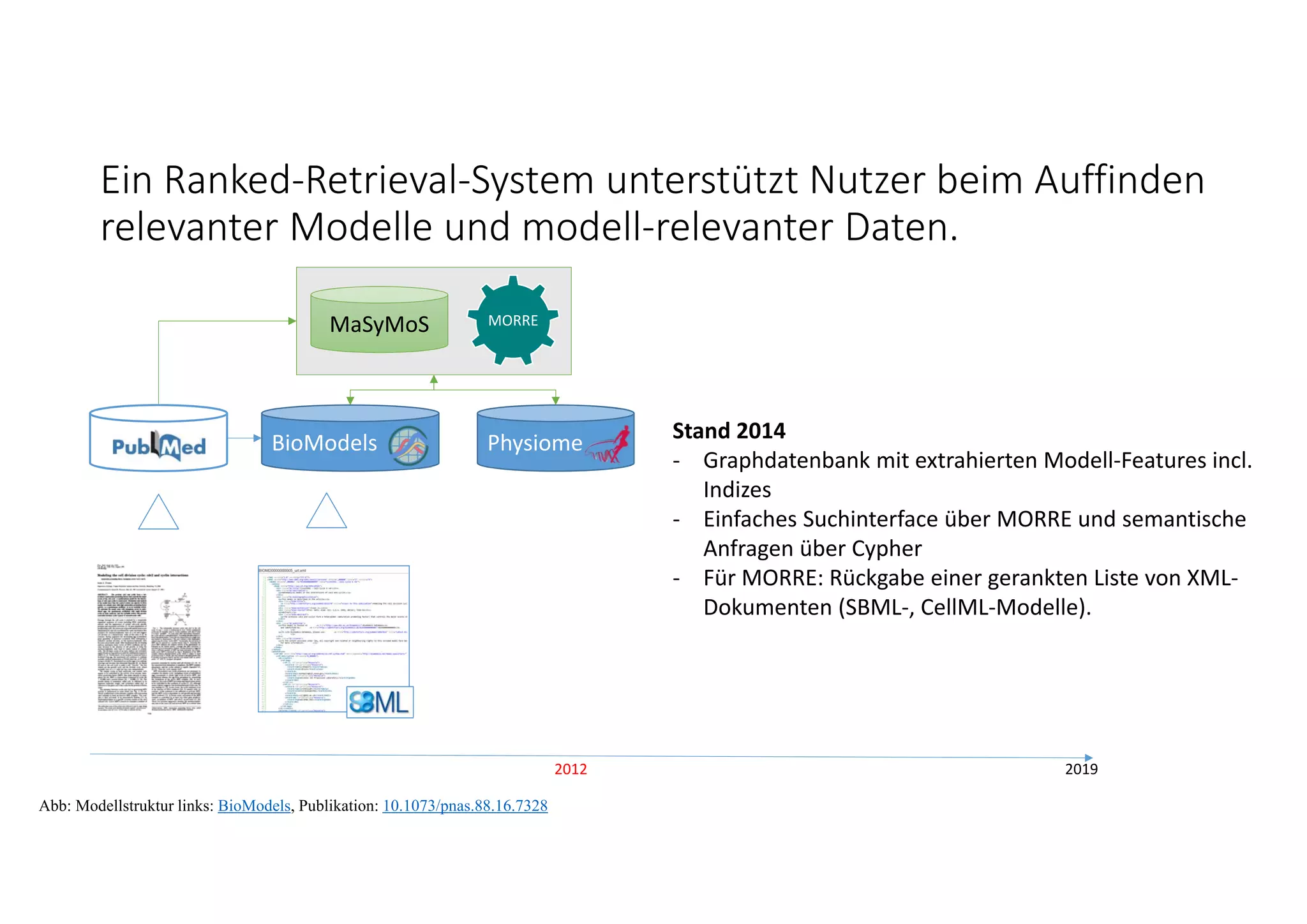
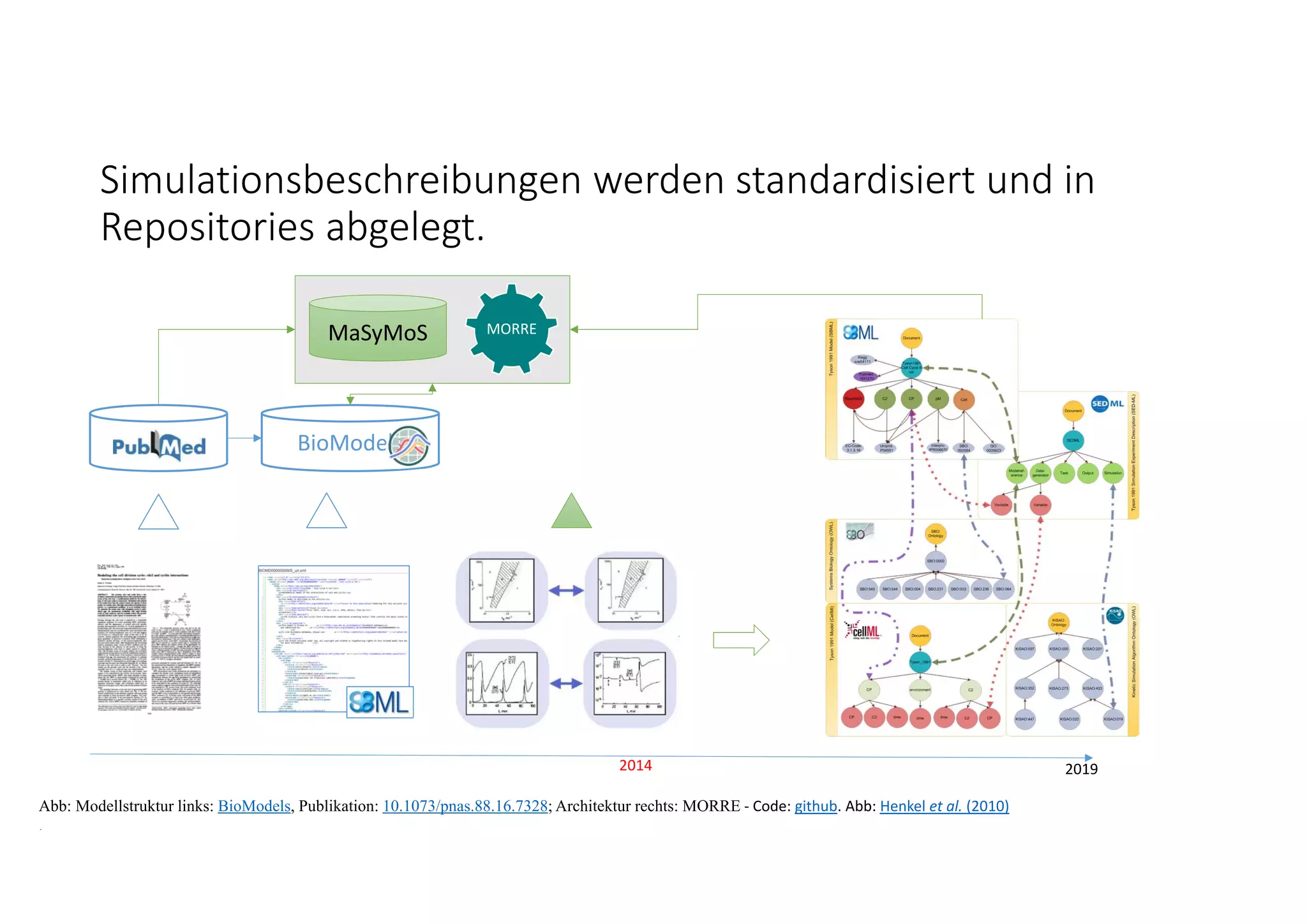
Abb: Modellstruktur links: BioModels, Publikation: 10.1073/pnas.88.16.7328; Rechts: Beispiel Struktursuche auf Modellen, R. Henkel, Code: Github. Abb: Oxford Lambusch et al (2018)
.
MATCH (n:SBML_MODEL)‐‐>(s1:SBML_SPECIES)‐[:IS_REACTANT]‐>(r1:SBML_REACTION) ‐
[:HAS_PRODUCT]‐>(s2:SBML_SPECIES)‐[:IS_REACTANT]‐>(r2:SBML_REACTION)
‐[:HAS_PRODUCT]‐>(s1)
RETURN n.NAME as Model_Name, count(s1)/2 as Cycle order by Cycle desc
<species id="C_p" sboTerm="SBO:0000247">
<annotation>
<rdf:Description rdf:about="C_p">
<bqbiol:is>
<rdf:Bag>
<rdf:li rdf:resource=
"urn:miriam:obo.chebi:CHEBI%3A27732"/>
</rdf:Bag>
</bqbiol:is>
<bqbiol:is>
<rdf:Bag>
<rdf:li rdf:resource=
"urn:miriam:kegg.compound:C07481"/>
</rdf:Bag>
</bqbiol:is>
</rdf:Description>
</annotation>
</species>
Model_Name Cycle
Stanford2013 ‐ Kinetic model of yeast metabolic network (standard) 2144
Stanford2013 ‐ Kinetic model of yeast metabolic network (regulation) 2144
Proctor2010 ‐ UCHL1 Protein Aggregation 1827
Smallbone2013 ‐ Yeast metabolic model with modular rate law, merged with Pritchard 2002 790
Smallbone2013 ‐ E.coli metabolic model with linlog rate law 689
Smallbone2013 ‐ Yeast metabolic model with modular rate law 681
Smallbone2013 ‐ Yeast metabolic model with linlog rate law 681
… …
Tyson1991 ‐ Cell Cycle 6 var 3
MaSyMoS MORRE
BioModels](https://image.slidesharecdn.com/waltemath-henkel-19-05-10-germanbiobank-190511201744/75/Implementierung-Graph-basierter-Ansatze-fur-das-Management-systembiologischer-Simulationsmodelle-21-2048.jpg)