9.traduccionpregrado
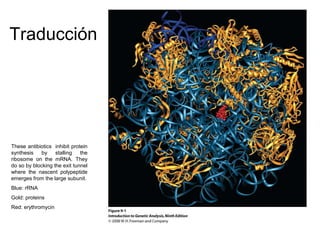
- 1. Traducción These antibiotics inhibit protein synthesis by stalling the ribosome on the mRNA. They do so by blocking the exit tunnel where the nascent polypeptide emerges from the large subunit. Blue: rRNA Gold: proteins Red: erythromycin
- 2. Colinearidad entre un gen y una proteína • La hipótesis de: un gen---un polipéptido de Beadle y Tatum fue la primera fuente de explicación de la función de un gen. • Los genes son de alguna manera responsables de la función de la enzimas y cada gen aparentemente controla una enzima • En 1953 con la estructura del DNA se supone de la colinearidad entre un gen y una proteína.
- 3. • En 1963, Yanofsky de la Universidad de Stanford, demostró la colinearidad entre la secuencia de los aminoácidos de una proteína y la secuencia de nucleótidos – Probó la relación entre genes alterados y proteínas alteradas • Aisló 16 mutaciones del gen trpA (síntesis de triptofano, subunit) todas formas inactivas de la enzima – Cada una resultó en la substitución de un aminoácido en diferentes posiciones en la proteína – Los sitios mutacionales en el mapa genético (x recombinación) del gen trpA aparecieron en el mismo orden que los correspondientes aminoácidos en la cadena polipeptídica)
- 4. Mutations in trpA are colinear with the amino acid changes. Although the order of the mutations on the gene map and the aminoacid positions are the same, the relative positions differ because the gene map is derived from recombination frequencies, which are not uniform along the length of the gene
- 5. • Cómo el DNA dicta la secuencia de una proteína – Si genes son segmentos de DNA y si el DNA es una cadena de nucleótidos, entonces las secuencia de nucleótidos dicta la secuencia de aminoácidos en una proteína – Código formado por nucleótidos – La combinación de letras de estos determinará la formación de palabras que representan los diferentes aminoácidos – Cuántas letras en el mRNA hacen una palabra o Codón • Es el código genético sobrepuesto o no • Cuántas letras se necesitan para todos los aminoácido
- 6. Código sobrepuesto o nosobrepuesto • Para un código nosobrepuesto, aminoácidos consecutivos son específicados por codones (código de palabras) consecutivos • Para un código sobrepuesto, aminoácidos consecutivos son especificados por codones que tienen en común algunas bases consecutivas en el siguiente codón • 1961 se sabía que el código no es sobrepuesto
- 7. Figure 10-24. The difference between an overlapping and a nonoverlapping code. The case illustrated is for a code with three letters (a triplet code). An overlapping code uses codons that employ some of the same nucleotides as those of other codons for the translation of a single protein, as shown at the top of the diagram (for the mRNA sequence shown at the bottom of the diagram). In a nonoverlapping code, a protein is translated by reading codons that do not share any of the same nucleotides. Note that the designation of an amino acid as aa2, aa3, and so forth, in the nonoverlapping model does not mean it is necessarily the same amino acid as its numerical counterpart in the overlapping model. The reason is that the triplets making up the respective codons for the two aa3's, for example, are different and would more than likely encode different amino acids. Identical amino acid numbers between the two models merely indicate the same amino acid position on the protein chain.
- 8. Número de letras en el codón • Si un mRNA es leído de un término al otro, sólo uno de los cuatro nucleótidos puede ser encontrado en cada posición. • Así si las palabras que codifican aminoácidos son de una sola letra, solo 4 palabras serían posibles, este vocabulario no puede ser entonces el código genético que sabemos que esta formado de 20 aminoácidos • Si las palabras (codones) son de dos letras, solo 4x4= 16 palabras son posible, ej. AU, CU o CC. Este vocabulario todavía es muy corto • Si las palabras son de tres letras entonces 4X4X4= 64 palabras son posibles, este vocabulario provee de más que suficiente palabras para describir los aminoácidos
- 9. Uso de supresores para demostrar el triplete de codones • Prueba experimental del triplete de codones – 1961 Francis Crick, Sidney Brenner y colaboradores • Mutantes del locus rII (gen que controla la lisis) del fago T4. • T4 tiene normalmente dos hospederos cepa tipo B y K de E. coli. En la mutación en rII sólo crecen fagos en la cepa tipo B pero no K.
- 10. • Las mutaciones fueron inducidas al utilizar un químico llamado proflavina que actúa por simple adición o deleción de un solo nucleótido –En una mutación llamada FCO, se encontraron reversiones que fueron detectadas porque producen placas tipo silvestres. » no eran idénticas al tipo silvestre ya que la reversión no se dió por la reversión de la mutacion original y porque que fueron capaces de crecer en E. coli tipo K
- 11. »La reversión se produjo por una segunda mutación encontrada en el mismo gen pero en una posición diferente. Esta mutación suprimió el efecto de la mutación FCO y por lo tanto se denomina: supresor (suppresor mutation) »Esta mutacion puede ser separada de la original por recombinacion
- 12. Figure 10-25. The suppressor of an initial rII mutation is shown to be an rII mutation itself after separation by crossing over. The original mutant, FCO, was induced by proflavin. Later, when the FCO strain was treated with proflavin again, a revertant was found, which on first appearance seemed to be wild type. However, a second mutation within the rII region was found to have been induced, and the double mutant rIIx rII y was shown not to be quite identical with the original wild type.
- 13. • Cómo estos resultados pueden ser explicados? – Si asumimos que la lectura del código es polarizada es decir que el gen es leído desde un extremo, – y que cada aminoácido es especificado por un triplete de bases, entonces la mutación original producida por proflavina (inserción o deleción) – interrumpió el marco de lectura original (open reading frame, ORF) (frameshift mutation) – y puede ser restablecida por una segunda inserción o deleción que compensa la primera mutación
- 14. • Frame shift mutation –THE FAT CAT EAT THE BIG RAT –Deletion of C: »THE FAT ATA TET HEB IGR AT –Insertion of A »THE FAT ATA ATE THE BIG RAT, la inserción suprime el efecto de la deleción
- 15. • Si asumimos que el mutante FCO es causado por una inserción, entonces la segunda mutación (supresor) podría ser una deleción puesto que restablece el marco de lectura abierto – Mensaje tipo silvestre » CAU CAU CAU CAU CAU – Mutación rlla: adición » CAU ACA UCA UCA UCA U – Mutación rIIa y rIIb (deleción): Pocas palabras son incorrectas pero el ORF es restablecido para las últimas letras » CAU ACA UCU CAU CAU
- 16. Degeneración del Código Genético • Se estableció 64 posible codones, palabras o tripletes. • Con sólo 20 palabras necesarias para los 20 aminoácidos para qué son utilizadas el resto de las palabras o codones? • El trabajo de Crick sugiere que el código genético es degenerado, lo que significa que todos los 64 tripletes deben tener un significado dentro del código. – Para que el código sea degenerado, algunos aminoácidos deben ser especificados por al menos dos o más diferentes tripletes – La razón para esto es que si solo se utilizan 20 tripletes el resto (44) no tendrían sentido si no codificaran ningún aminoácido. En este caso la mayoría de las mutaciones (frameshift mutations) producirían palabras sin sentido lo que detendría el proceso de síntesis de proteínas y la supresión raramente trabajaría.
- 17. Determinación del código • Diversos experimentos han demostrado que triplete específica un determinado aminoácido – Sintesis in vitro de mRNA con la enzima polinucleótido fosforilasa: Sólo se requieren los ribonucleótidos y una cadena simple de RNA es sintetizada. No se requiere de una cadena molde en esta reacción • El primero fue obtenido al mezclar sólo uracilo (poliU) • En 1961 Nirenberg y Matthaei mezclaron este poliU con la maquinaria sintetizadora de E. coli in vitro y observaron la formación de una proteína.. El aminoácido incorporado fue fenilalanina. Recibieron el Premio Nobel • De este experimento muchos códigos fueron descifrados – H. Korana recibió el premio Nobel al descifrar el código genético utilizando mRNA que contenían dos tipos diferentes de nucleótidos (AGA)n.
- 22. Codones de terminación • Codones de terminación (STOP codons): Codones sin sentido:(nonsense codons) TAG UAG amber TGA UGA opal TAA UAA ochre
- 24. • http://bcs.whfreeman.com/webpub/Ektron/ Hillis Principles of Life2e/Animated Tutorials/pol2e_at_1003_Deciphering_the _Genetic_Code/pol2e_at_1003_Decipheri ng_the_Genetic_Code.html • https://www.dnalc.org/view/16031- Marshall-Nirenberg-1961.html
- 25. • Reconocimiento del codón por el tRNA – Son adaptadores que reconocen los codones en el mRNA e insertan apropiadamente su aminoácido • Anticodón • Sitio de adhesión del aminoácido • Los otros brazos probablemente asisten al enlace del tRNA al ribosoma • Bases nitrogenadas no comunes – Pseudouridina (), metilguanosina (mG), dimetilguanosina (m2G), metilinosina (mI) y dihidrouridina (DHU) • Cada uno tiene una estructura tridimensional única que le permite su reconocimiento adecuado por la sintetaza – En forma de L, que le confiere máxima estabilidad • Se localizan su genes nuclearmente (tandem repeats)
- 28. Figure 10-26. The structure of transfer RNA. (a) The functional areas of a generalized tRNA molecule. (b) The specific sequence of yeast alanine tRNA. Arrows indicate several kinds of rare modified bases. (c) Diagram of the actual three-dimensional structure of yeast phenylalanine tRNA. The abbreviations y, mG, m2G, mI, and DHU (or UH2) are abbreviations for modified bases pseudouridine, methylguanosine, dimethylguanosine, methylinosine, and dihydrouridine, respectively. (Part a from S. Arnott, "The Structure of Transfer RNA," Progress in Biophysics and Molecular Biology 22, 1971, 186; parts b and c from L. Stryer, Biochemistry, 4th ed. Copyright © 1995 by Lubert Stryer; part c based on a drawing by Sung-Hou Kim.)
- 29. When folded into their correct three dimensional structures, the yeast tRNA for glutamine (blue) almost completely overlaps the yeast tRNA for phenylalanine (red) except for the anticodon loop and the aminoacyl end Aunque los tRNA se diferencian en la secuencia de nucleótidos, todos ellos se pliegan en la misma conformación L, la conservación de la estructura sugiere que la forma es importante para la función
- 30. • Múltiples codones para un sólo aminoácido – Degeneración del código genético: 1 para trp y 6 para Ser – Varias explicaciones • Ciertos aminoácidos pueden ser traídos al ribosoma por medio de diferentes tRNA con diferentes anticodones • Ciertos tipos de tRNA cargados pueden llevar su aminoácido específico a cualesquiera de varios codones relacionados no solamente uno (el complementario), por medio de un apareamiento no especifico en el final (3’ en el codon y 5’ en el anticodon),: Bamboleo, Tambaleo (Wobble)
- 31. Figure 10-28. In the third site (5 end) of the anticodon, G can take either of two wobble positions, thus being able to pair with either U or C. This ability means that a single tRNA species carrying an amino acid (in this case, serine) can recognize two codons UCU and UCC in the mRNA.
- 33. Tres tRNAs diferentes que pueden aparear con codones para serina. Algunos organismos tiene un cuarto tRNA que tiene un anticodón idéntico con uno de los aquí mostrados pero se diferencia en su secuencia de nucléotidos en alguna parte de la molécula; son llamados isoaccepting tRNAs porque aceptan el mismo aminoácido pero son transcritos de diferentes genes de tRNA
- 34. Síntesis de Proteínas • Cómo una reacción química: – Cada aminoácido es adherido a una molécula de tRNA específica mediante un enlace rico en energía derivado del ATP. Catalizado por una sintetasa (aminoacyl-tRNA sinthetase): • aa1+ tRNA1+ATP sintetaza aa1---tRNA + AMP+ Ppi • Hay una sintetasa para cada aminoácido (20) • El producto es un tRNA cargado – La energía de este tRNA es convertido en un enlace peptídico mediante el enlace de otro aminoácido en el ribosoma • aa1—tRNA1 + aa2—tRNA2 peptidil transferasa en el ribosoma aa1—aa2—tRNA2 + tRNA1 liberado, nuevos aminoácidos son añadidos mediante la formación de enlace peptídico en la cadena polinucleótido
- 37. Ribosomas • La síntesis de proteínas se lleva a cabo cuando el tRNA y el mRNA se asocian con los ribosomas • La tarea del tRNA y el ribosoma es traducir la secuencia de codones en el mRNA en secuencias de aminoácidos en la proteína
- 38. Figure 10-30. A ribosome contains a large and a small subunit. Each subunit contains both rRNA of varying lengths and a set of proteins (designated by different shapes and shading). There are two principal rRNA molecules in all ribosomes. (a) Ribosomes from prokaryotes also contain one 120-base-long rRNA that sediments at 5S. (b) Eukaryotic ribosomes have two small rRNAs: a 5S RNA molecule similar to the prokaryotic 5S, and a 5.8S molecule 160 bases long. The large subunit proteins are named L1, L2, and so forth, and the small subunit proteins are named S1, S2, and so forth. (From H. Lodish, D. Baltimore, A. Berk, S. L. Zipursky, P. Matsudaira, and J. Darnell, Molecular Cell Biology, 3d ed. Copyright © 1995 by Scientific American Books, Inc.) S: coeficiente de Svedberg, indicación del tamaño molecular Ribosomas
- 40. •Aunque los ribosomas eucarióticos son más grandes debido a un mayor número de componentes; los componentes y los pasos en la síntesis de proteínas son similares. Esto indica que este proceso se originó en un ancestro común de procariotas y eucariotas. •El rRNA se piensa lleva a cabo los pasos importantes de la síntesis de proteínas asistido por las proteínas ribosomales The folded structure of the prokaryotic 16S ribosomal of the small ribosomal subunit
- 41. • En el esquema general de la síntesis: – el mRNA enlaza la subunidad pequeña de los ribosomas. – El tRNA enlaza en tres sitios en el ribosoma • Sitio A (aminoacyl): Sitio de entrada del aminoacil-tRNA • Sitio P (Peptidyl): que lleva el creciente polipéptido • Sitio E (Exit): tRNA deacetilado (no lleva un aminoácido)
- 42. – Decoding center: En la subunidad pequeña, se asegura que solo los tRNAs con el anticodon correcto (cognate tRNAs, cognado) se apareen con el codón y sean aceptados en el sitioA – Peptidyl transferase center: en la 50S, el tRNA apropiado se asocia donde se cataliza la formación del enlace peptídico. – Ambos centros están formados de regiones de rRNA, son sitios importantes de contactos de tRNA-rRNA – El enlace peptídico puede ser catalizado por un sitio activo en el rRNA y solo asistido por proteínas ribosomales. Así 50S puede funcionar como una Ribozima – Etapas: • Iniciación, elongación y terminación
- 43. Figure 10-31. The addition of a single amino acid to the growing polypeptide chain in the course of translation of mRNA.
- 45. • Iniciación – IF1, IF2, IF3 – En procariotas un tRNA iniciador que incorpora formilmetionina: tRNAfMET – Codones de iniciación en procariotas: ATG (AUG), GTG (GUG) y en raras ocasiones TTG (UUG) – Los ribosomas existen como subunidades libres en el citosol hasta que se inicie la síntesis de proteínas
- 46. Figure 10-32. The structures of methionine (Met) and N-formylmethionine (fMet). A tRNA bearing fMet can initiate a polypeptide chain in prokaryotes but cannot be inserted in a growing chain; a tRNA bearing Met can be inserted in a growing chain but will not initiate a new chain. Both these tRNAs bear the same anticodon complementing the codon AUG.
- 47. Figure 10-35. Binding of the Shine-Dalgarno sequence on an mRNA to the 3 end of 16S rRNA. (After L. Stryer, Biochemistry, 4th ed. Copyright © 1995 by Lubert Stryer.)
- 48. Este apareamiento correctamente posiciona el codón de iniciación en el sitio P donde el tRNA iniciador se va a enlazar
- 49. IF3: 30S separada de 50S IF1 e IF2: sólo el tRNA iniciador enlace al sitio P
- 51. Figure 10-33. Steps in the initiation of translation (see text).
- 52. • Iniciación en eucariotas: – Transcripción y traducción en compartimentos diferentes – En el citoplasma mRNA es cubierto por proteínas y las regiones pueden ser helicales dobles debido al apereamiento intramolecular de las bases. Estas regiones de estructuras secundarias deben ser removidas para exponer el codón de iniciación ATG • Es llevado a cabo por eIF4A, B y G. Se asocian con el cap, con la subunidad 40S y el iniciador tRNA para formar un complejo de iniciación. Una vez en su lugar el complejo se mueve en la dirección 5’---3’ para desdoblar la estructura secundaria y al mismo tiempo para buscar la secuencia AUG donde se iniciará la traducción
- 56. Figure 10-36. Steps in elongation (see text).
- 57. Figure 10-37. Steps leading to termination of protein synthesis (see text).
- 58. RF1:UAA, UAG RF2: UAA, UGA RF3 asiste a ambos
- 61. El Proteoma • Genoma: Completo material genético de un juego de cromosomas • Transcriptoma: Colección completa de secuencias transcritas en el genoma • Proteoma: Juego completo de proteínas que puede ser expresada por el material genético de un organismo – Puede ser enriquecido por dos procesos celulares: el empalme alternativo y modificaciones postraduccionales
- 62. • El corte y empalme alternativo genera proteínas isoformas con diferentes combinaciones de dominios funcionales
- 63. • Eventos postraduccionales: – El plegamiento de una proteína dentro de la célula: • Proteínas nativas vs no nativas • El ambiente acuoso de la célula no favorece al plegamiento correcto de las proteínas – Chaperonas: » Plantas, animales, bacterias » GroE chaperoninas:Complejo de múltiples subunidades llamadas máquinas chaperoninas de plegamiento » No se conoce el mecanismo exacto entran en una cámara en la máquina de plegamiento que proporciona un microambiente eléctricamente neutro dentro del cual las proteínas pueden adoptar su conformación nativa
- 64. – Modificaciones postraduccionales de las cadenas laterales de aminoácidos • Más comunes fosforilación y ubiquitinación • Fosforilación: – quinasas: Ser, thr, tyr – Fosfatasas – Cambio de conformación y funciona como un interruptor reversible para controlar actividades: enzimáticas, interacciones prot-prot, DNA-prot – La mayoría de interacciones entre proteínas en la célula esta regulada por fosforilación • INTERACTOMA: conjunto completo de interacciones entre proteínas
- 66. Proteins represented by circles interact with other proteins to form simple or large protein complexes Todas las interacciones entre proteínas en un organismo componen el interactoma
- 68. • Ubiquitinación: adición a la porción amino de los residuos de lisina de múltiples copias de ubiquitina – 76a.a. sólo en eucariotas – La marca para la degración de la proteasa: 26S Proteosoma • Proteínas de vida corta como las reguladoras del ciclo celular y las que están dañadas o mutadas • Destino de las proteínas
