PCR Reação em Cadeia
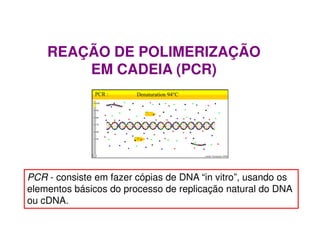
- 1. REAÇÃO DE POLIMERIZAÇÃO EM CADEIA (PCR) PCR - consiste em fazer cópias de DNA “in vitro”, usando os elementos básicos do processo de replicação natural do DNA ou cDNA.
- 2. O processo de PCR foi descrito por Kary Mullis no final da década de 1980, tendo-lhe sido posteriormente, em 1993, atribuído o Prêmio Nobel de Química pelo seu trabalho.
- 3. Em 1989, a Hoffman La Roche & Perkin-Elmer Corporation patentearam este processo. Bactéria Thermus aquaticus (Taq-polimerase).
- 4. PRINCÍPIOS DA REAÇÃO DE PCR A reação de polimerização em cadeia (“Polimerase chain reaction”, PCR) é a técnica que permite a amplificação do DNA ou cDNA in vitro, utilizando-se basicamente de uma reação enzimática catalizada pela polimerase (enzima termoestável) cuja atividade depende de ions Mg++ Ocorre em 3 etapas:Ocorre em 3 etapas: a) “Melting”(ou desnaturação que consiste na separação da dupla fita do DNA a ser amplificado), b) “annealing”( anelamento ou hibridização- ligação do iniciador ou “primer” ao DNA a ser amplificado), c) “extension” (extensão ou seja a polimerização propriamente dita)
- 5. Depende: Alternância de temperaturas a cada ciclo: Replicação in vitro do DNA • DESNATURAÇÃO • ANELAMENTO • POLIMERIZAÇÃO
- 6. Alternância de temperaturas a cada ciclo 1:Denaturaçao 94 ° C -96 ° C Separa a fita dupla do DNA molde em duas fitas simples 2.Anelamento: 37 ° C -65 ° C37 ° C -65 ° C Primers se ligam aos sitios específicos na fita simples 3. Extensão: 72 ° DNA polymerase acrescenta os dNTPs à fita, de acordo com o pareamneto de bases
- 7. Inicialmente é necessário que as duas fitas de DNA a ser amplificado sejam separadas. A elevação da temperatura entre 90 a 95 ºC promove a separação da fita dupla de DNA em duas fitas simples. Muitas vezes a temperatura de desnaturação é determinada empiricamente e depende de alguns fatores como a quantidade de guanina e citosina (GC) do fragmento DNA alvo ou de interesse. a)Desnaturação
- 8. A polimerase para anexar as bases complementares, transformando as fitas simples em fitas duplas, necessita de um fragmento de DNA já ligado na região previamente escolhida. Para isso adicionam-se, à solução, os “primers” ou iniciadores que são pequenos fragmentos sintéticos de DNA de fita simples, oligonucleotídeos de 20 a 30 bases nitrogenadas de comprimento, que são sintetizados in vitro baseado na sequência do DNA a ser amplificado sendo eles complementares à seqüência do segmento de DNA de interesse. Nas duas fitas surgem, assim, um pequeno fragmento de DNA de dupla fita intacta. A hibridização dos “primers” descrito como “Annealing” (do inglês), deve ser feito a uma tempertura inferior à da desnaturação (45 a 70 °°°°C). b) Anelamento-
- 9. - Quando os “primers” encontram e ligam- se aos segmentos complementares, a DNA-Polimerase liga os nucleotídeos entre si, completando assim as fitas simples e tornando-as duplas (extensão). A DNA- Polimerase não reconhece apenas o bloco de início como também consegue diferenciar as duas extremidades dos “primers” (3’ e 5’). Ela inicia a reação sempre pela extremidade 3’ do “primer”, completando a c) Extensão fita simples daí em diante, portanto, sempre na direção do fragmento procurado.
- 12. Automação da PCR Um aparelho de PCR, ou um termociclador, repete o correspondenteUm aparelho de PCR, ou um termociclador, repete o correspondente a programa de temperaturas. O operador deve apenas programar e dar início para que transcorra a amplificação Técnicamente muitas mudanças tem sido introduzidas melhorando cada vez mais e ampliando a sua utilização. Para cada situação experimental, novas modificações têm de ser introduzidas, de tal forma que é muito difícil descrever um único procedimento para um uso generalizado. Por outro lado, é fundamental o conhecimento básico das etapas envolvidas na técnica, para um desenho experimental com sucesso
- 13. É um seguimento de DNA ou RNA de 15 a 40 bases, geralmente baseado na sequência já conhecida do ácido nucleico, sintetizado quimicamente por instrumentos automáticos, que podem ser adquiridos no comércio para fins já definidos ou se escolhe a região desejada do DNA ou RNA a ser amplificada. Primer: É um segmento de DNA ou RNA que foi marcado seja com enzimas,, substratos antigenicos, substâncias quimioluminescentes ou radioativas, que podem ligar-se com alta especificidade a uma sequência complementar do ácido nucleico alvo (escolhido). Sonda (probe):
- 14. EFICIÊNCIA NA AMPLIFICAÇÃO: Modelo teórico eficiência do processo de amplificação corresponde a 100%, o que não é observado na prática. Experimentalmente de 78% a 97%, dependendo do fragmento amplificado. Os principais problemas para a padronização: a) Não detecção do produto, b) Baixo rendimento do produto desejado, c) Presença de bandas não específicas devido a “misextension” dos “primers”, d) Formação de dímeros dos “primers” que competem pela amplificação com o produto desejado, mutação ou heterogeneicidade devido a erro de incorporação.
- 15. Após cada ciclo, o número de fragmentos se duplica: de um formando-se 2, e então novamente 4, 8, 16, 32, 64, 128... de tal forma que pode ser definida matematicamente por: PCR REAÇÃO EM CADEIA de tal forma que pode ser definida matematicamente por: N = No ×××× 2n N = número de moléculas amplificadas No = número de moléculas iniciais n = número de ciclos de amplificação
- 16. Primeiro Ciclo da PCR
- 18. A polimerização é feita por uma DNA polimerase termoestável = síntese exponencial de moléculas de DNA. São utilizados dois primers que delimitam o segmento amplificado
- 19. Cada uma das novas fitas de DNA extendidas a partir dos primers serve como molde para a síntese de uma nova fita. Isso garante o aumento exponencial do número de novas fitas Todas as fitas de DNA sintetizadas durante a PCR tem a sequência dos primers na extremidade 5’ e atem a sequência dos primers na extremidade 5’ e a sequência complementar aos primers na posição 3’.
- 20. Polimerização 5’ 3’ Todas as DNA-polimerasesTodas as DNA-polimerases requerem para seu funcionamento: Molécula de DNA molde Primer de oligonucleotídeos Magnésio dNTPs
- 21. Qualquer sequência alvo de DNA pode ser amplificada por PCR •A localização da sequencia-alvo é feita pelos primers. • Oligonucleotídeos com 20-24 pares de bases são seletivos o suficiente para localizar um sítio único em um genoma de alta complexidade. • O pareamento dos primers com a sequencia –alvo se faz pelo estabelecimento de pontes de hidrgênio, segundo Watson e Crick
- 23. • Tampão:Mantém o pH ótimo para a atividade da enzima • Sais: fornecem condições ótimas para a máxima eficiência da enzima. influenciam na cinetica de hibridizaçao do DNA ou na Tm dos primers. Componentes da PCR • Magnésio (Mg++): ions de Mg são necessários para estimular a Taq polimerase Mg++: 1 a 4 mM
- 24. Cinética da PCR •Fase de screening- ciclos iniciais Os primers procuram o molde de DNA com as sequencias que lhes são complementares . Encontrar a sequencia complementar não é dificil, pois os primers estão em considerável excesso em relação ao molde. • Fase intermediária: O processo de amplificação está ocorrendo levando ao acúmuloO processo de amplificação está ocorrendo levando ao acúmulo exponencial do fragmento de DNA.O pareamento do primer com a sequencia ja está facilitada, pois ja existem várias cópias da sequencia-alvo • Fase tardia ou fase de platô: A amplificaçào ja é sub-ótima devido à limitaçao dos reagentes e a competição dos produtos gerados com os primers disponíveis.
- 26. O aumento exponencial do no de cópias ocorre até um determinado no de ciclos, que será variavel de reação para reação. Causas do platô (Desvio do ideal teórico): • Perda de atividade da enzima • Todas as enzimas disponíveis estão ocupadas com a síntese de DNA • Acúmulo de produtos amplificados que tendem a parear entre si,• Acúmulo de produtos amplificados que tendem a parear entre si, em detrimento do pareamento com os “primers”. Aumenta possibilidade de amplificação de produtos inespecíficos. • Gasto dos reagentes, especialmente de primers, mas também de deoxinucleotídeos. • Acúmulo de pirofosfato, resultante das ligações fosfodiésteres. • Competição com outros produtos que vinham sendo amplificados com eficiência menor mas também foram se acumulando.
- 27. Fidelidade na PCR • Fidelidade: Acuracia na replicação •Fidelidade na PCR Alto % dos produtos da PCR tem seqüências idênticas ao alvoAlto % dos produtos da PCR tem seqüências idênticas ao alvo • Alta fidelidade
- 28. Condições que afetam a fidelidade da PCR Fatores Aumenta Fidelidade Reduz fidelidade dNTPs Balanceado 40-50µM de cada Desbalanceado 1-2mM de cada Mg++ 1:1 com os dNTPs 6-8 mM Mg++ Temp. anelamento Enzima ou tempo de extensão Ciclos
- 29. Otimização da PCR • Aumenta a eficiência da reação (produção) • Aumenta a especificidade • Melhora a reprodutibilidade Parâmetros a serem considerados na otimização • Desenho de primers Conteúdo de GC, Evitar longas regiões ricas em GC ou AT, Temperatura de anelamento (Tm – 4oC) • Condições dos ciclos • Concentração dos reagentes •Componentes do tampão
- 30. Parâmetros dos ciclos: 1. Temperatura e Tempo de desnaturação • Falta de desnaturação do molde é uma causa comum de falha na PCRcomum de falha na PCR • Tipicamente 94oC por 30 s • Tempo de desnaturação muito longo reduz a meia vida da enzima
- 31. Parâmetros dos ciclos: 2. Temperatura e Tempo de anelamento • Temperatura de anelamento = 5oC abaixo do Tm real dos primersdos primers • Tempo de anelamento = 15 a 30 s • Tempo de extensão: função tamanho do AMPLICON – 1 minuto é suficiente 1,2 kb – Longo, reduz a meia vida da enzima
- 32. DNA Polimerases Termoestáveis Thermos aquaticus (Taq polymerase) • Taxa de incorporação de bases erradas pode variar entre 10-3 a 10-6, dependendo das concentrações de Mg2+ e dNTPs • A incorporação de base errada (mismatch) reduz a estabilidade da interação da enzima com o DNA, favorecendo a interrupção da polimerização. • Baixa temperatura de extensão e elevada concentração de dNTPs favorecem a extensão de primers com pareamento imperfeito e a extensão após mismatch.
- 36. Inibem a PCR •Contaminantes da amostra de DNA (limitante) Inibidores da Polimerase •Fenol •Proteinase K•Proteinase K •Agentes quelantes em excesso (EDTA) •SDS •Elevadas concentrações de sais
